BIOはOpenSSLにおけるフレームワークにおける抽象データ型で,暗号化や符号化,ファイルやネットワークといった入出力の詳細をアプリケーションから隠蔽する.で,このフレームワークのもとで実装されているBase64コーデックを試してみた.
BIOって何だ?
base64エンコードするサンプル
base64デコードするサンプル
サンプルの実行
BIOって何だ?
BIOはOpenSSLにおける入出力関連のフレームワークにおける抽象データ型である. BIOを使用することにより,アプリケーションでは変更不要なある「お決まりの形式」でプログラミングしつつ,容易にファイル/ネットワークとか,暗号化形式や符号化形式を切替えることができる. さらに,BIOはチェインを組むことができ,例えば暗号化して符号化してネットワークに出力するというBIOのチェイン(このチェイン自信もBIO型となる)を作成して書き出すのも,データをそのままファイルに書き出すのも同様の記述によって表現できるようになる.
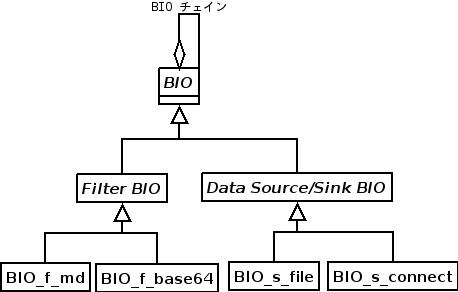
上の図はBIOのクラス図である. BIO_f_md,BIO_f_base64,BIO_s_file,BIO_s_connectは実際には関数だが,イメージを掴みやすくするために,上のように記述した. man bioによると,BIOには二種類ある.
ソース/シンクBIO
ソケットBIOとか,ファイルBIOといった,データのソース(源),シンク(宛先)を実装したBIO.
フィルターBIO
暗号化など,他のBIOからデータを受け取り,別のBIOにデータを渡すフィルターの役割を果たす. フィルターBIOに格納されているデータが変更されるかどうかは,BIOの実装やオペレーションによる.
base64エンコードするサンプル
では,実際にBIOを使用したプログラミングを試してみる. 今回はbase64でエンコードするサンプルとデコードするサンプルを作成してみる. まずはエンコード.
#include <stdio.h>
#include <string.h>
#include <openssl/bio.h>
#include <openssl/evp.h>
int
main(int argc, char *argv[])
{
if (argc != 3)
{
fprintf(stderr, "Usage : %s outfile message\n", argv[0]);
exit(1);
}
// 出力BIOの作成
BIO *bioOutFile = BIO_new_file(argv[1], "w");
if (bioOutFile == NULL)
{
perror("failed to BIO_new_file for bioOutFile.");
exit(1);
}
// フィルターBIOの作成
BIO *bioBase64 = BIO_new(BIO_f_base64());
// BIOのチェイン
BIO *chainBio = BIO_push(bioBase64, bioOutFile);
// 符号化およびファイル出力
int len = BIO_write(chainBio, argv[2], strlen(argv[2]));
if (len >= 0)
{
fprintf(stdout, "wrote %d bytes\n", len);
}
else
{
fprintf(stderr, "failed to BIO_write\n");
exit(1);
}
if (BIO_flush(chainBio) != 1)
{
fprintf(stderr, "failed to BIO_flush\n");
exit(1);
}
BIO_free_all(chainBio);
return 0;
}
まずデータシンクBIOを作成する. BIOの作成にはBIOの実装毎に専用の作成関数がある. 今回はファイルに出力するので,ファイル実装のBIOを作成するので,
BIO *
BIO_new_file(const char *filename, const char *mode);
を使用する. 第一引数がファイル名,第二引数はファイルをオープンする際のモードでfopen時のものと同様である. 返り値は作成したBIOのポインタであり,失敗するとNULLを返す.
// 出力BIOの作成
BIO *bioOutFile = BIO_new_file(argv[1], "w");
if (bioOutFile == NULL)
{
perror("failed to BIO_new_file for bioOutFile.");
exit(1);
}
類似機能で,引数をFILE*とする関数もあるが,これはデコードのサンプルで使用してみた. 次にbase64をするためのBIOを作成する. これには
BIO *
BIO_new(BIO_METHOD *type);
BIO_METHOD *
BIO_f_base64(void);
の二つの関数を使用する. BIO_newの引数にはフィルターBIOの処理実装を指定する. 今回はbase64なので,BIO_f_base64にて処理実装を取得し,それをBIO_newに渡している. BIO_newは作成したBIOを返すが,失敗するとNULLを返す.
// フィルターBIOの作成
BIO *bioBase64 = BIO_new(BIO_f_base64());
このように,データソース/シンクのBIOは
BIO_s_xxx
というパターンの関数名とBIO*の返り値をもつ. そしてフィルターBIOの処理実装は
BIO_f_xxx
というパターンの関数名とBIO_METHOD *の返り値をもつ. フィルターBIOの場合は,このBIO_METHODを使用してBIO_newでBIOを作成する流れになる. 次に,作成した二つのBIOをつなげてチェインにする. これには
BIO *
BIO_push(BIO *b,BIO *append);
を使用する. 第一引数のBIOの後に第二引数のBIOがぶら下がる. 返り値は第一引数のBIO自身である. これにより,第一引数のBIOに対して書き込み処理すると,その処理結果が第二引数のBIOに渡され書き込み処理がされる. このようにして一回の操作でチェインを組んでいるBIOにて順次処理が行われる.
// BIOのチェイン
BIO *chainBio = BIO_push(bioBase64, bioOutFile);
サンプルでは,chainBIO(bioBase64と同値)に対して書き込み処理をすると,bioBase64にて符号化処理されたのち,bioOutFileに渡され,ファイルに書き出される. 書き込みには
int
BIO_write(BIO *b, const void *buf, int len);
を使用する. 第一引数は対象BIO,第二引数は書き出しデータのバッファ,第三引数は書き出しデータのサイズである. 返り値は書き出したデータ長で,0か-1なら書き出しデータなし,-2なら指定されたBIOが実装されていない.
int len = BIO_write(chainBio, argv[2], strlen(argv[2]));
if (len >= 0)
{
fprintf(stdout, "wrote %d bytes\n", len);
}
else
{
fprintf(stderr, "failed to BIO_write\n");
exit(1);
}
読み込みの場合はチェインを伝わる順番が逆になるが,これはデコードのサンプルにて確認できる. 書き込みの場合はやはりフラッシュ操作がある.
int
BIO_flush(BIO *b);
引数は対象BIO,返り値は1なら成功,0か-1なら失敗. BIO実装によってはこれを呼び出すと,それ移行データを書き出さなくなるらしい.
if (BIO_flush(chainBio) != 1)
{
fprintf(stderr, "failed to BIO_flush\n");
exit(1);
}
最後にBIOをクローズしてリソースを開放する.
void
BIO_free_all(BIO *a);
これを実行すると引数とそれにチェインされているずべてのBIOに対し,
int
BIO_free(BIO *a);
が実行される. 途中のチェインでBIO_freeに失敗しても構わずにすべてのBIOに対してクローズ処理が実行される. BIO_freeの返り値は1なら成功,0なら失敗.
BIO_free_all(chainBio);base64デコードするサンプル
次にデコードのサンプルを示す.
#include <stdio.h>
#include <string.h>
#include <openssl/bio.h>
#include <openssl/evp.h>
int
main(int argc, char *argv[])
{
if (argc != 2)
{
fprintf(stderr, "Usage : %s infile\n", argv[0]);
exit(1);
}
// 入力BIOの作成
FILE *inFile = fopen(argv[1], "r");
if (inFile == NULL)
{
perror("failed to fopen for inFile.");
exit(1);
}
BIO *bioInFile = BIO_new_fp(inFile, BIO_CLOSE);
if (bioInFile == NULL)
{
perror("failed to BIO_new_file for bioInFile.");
exit(1);
}
// フィルターBIOの作成
BIO *bioBase64 = BIO_new(BIO_f_base64());
// BIOのチェイン
BIO *chainBio = BIO_push(bioBase64, bioInFile);
int len = 1;
char buf[512];
while((len = BIO_read(chainBio, buf, 512)) > 0)
{
fprintf(stdout, "%*s", len, buf);
}
fprintf(stdout, "\n");
BIO_free_all(chainBio);
return 0;
}流れは大体エンコードと同様. 今回はファイルBIOの作成に
BIO *
BIO_new_fp(FILE *stream, int flags);
を使用した. 第二引数にはたぶんBIO_CLOSEとBIO_NOCLOSEがあり,ライブラリにて勝手にファイルをクローズして欲しくない場合は後者を指定する.
// 入力BIOの作成
FILE *inFile = fopen(argv[1], "r");
if (inFile == NULL)
{
perror("failed to fopen for inFile.");
exit(1);
}
BIO *bioInFile = BIO_new_fp(inFile, BIO_CLOSE);
if (bioInFile == NULL)
{
perror("failed to BIO_new_file for bioInFile.");
exit(1);
}
あとはフィルターBIOを作成し,チェインを組んで読み込み.
// フィルターBIOの作成
BIO *bioBase64 = BIO_new(BIO_f_base64());
// BIOのチェイン
BIO *chainBio = BIO_push(bioBase64, bioInFile);
int len = 1;
char buf[512];
while((len = BIO_read(chainBio, buf, 512)) > 0)
{
fprintf(stdout, "%*s", len, buf);
}
fprintf(stdout, "\n");
チェインの順序がエンコード時と同じである点に注意. 読み込みの場合はチェインを逆からたどるらしく,これで正しく動作する. 読み込みには
int
BIO_read(BIO *b, void *buf, int len);
を使用する. 第一引数は対象BIO,第二引数は読み込みバッファ,第三引数は読み込みデータ長である. 返り値は読み込んだデータ長で,0か-1なら読み込みデータなし,-2は指定したBIO実装がない.サンプルの実行
では,上記二つのサンプルを実行してみる.
# hogeというファイルに文字列HOGEFUGAFOOVARをbase64エンコードして書き込む
$ ./myb64enc hoge HOGEFUGAFOOVAR
wrote 14 bytes
# hogeファイルの中身はbase64エンコードされたもの
$ cat hoge
SE9HRUZVR0FGT09WQVI=
# hogeファイルの中身をbase64デコードしてみる
$ ./myb64dec hoge
HOGEFUGAFOOVAR
